Identifizierung des dynamischen Bindens von Transkriptionsfaktoren mittels ATAC-seq footprinting
Forschungsbericht (importiert) 2020 - Max-Planck-Institut für Herz- und Lungenforschung - W. G. Kerckhoff-Institut
Transkriptionsfaktoren sind wichtig für die Steuerung von Zelltypen
Obgleich alle Zellen im menschlichen Körper die gleiche DNA-Sequenz enthalten, führen die verschiedenen Zelltypen jeweils ganz unterschiedliche Funktionen aus - ein Mechanismus, der als Differenzierung bezeichnet wird. Die Zell-Differenzierung wird durch die Expression bestimmter Gene gesteuert, die in der DNA-Sequenz selbst kodiert sind. Interessanterweise zirkuliert die DNA in Zellen nicht frei umher, sondern ist größtenteils um Proteinstrukturen namens Nukleosomen geschlungen. In diesem Zustand sind die Gene für den Ableseapparat der Zelle unzugänglich und damit inaktiv. Erst unter bestimmten Umständen lösen sich die Nukleosomen von der DNA und erlauben dadurch Proteinen, darunter vor allem den Transkriptionsfaktoren, an die Molekülkette zu binden und die Expression von Genen zu induzieren. Somit ist die Kontrolle der DNA-Zugänglichkeit entscheidend für die Steuerung des Aktivitätsstatus von Genen in einer Zelle.
Um herauszufinden, welche Stellen der DNA zugänglich sind, wurden in der Vergangenheit mehrere spezifische Tests entwickelt. Eine Labortechnik namens „ATAC-seq“ (Assay for Transposase-Accessible Chromatin using sequencing) macht sich ein Enzym namens Tn5-Transposase zunutze, das DNA gerne dort schneidet, wo sie zugänglich ist. An Stellen, an denen die DNA durch Nukleosomen geschützt ist, kann Tn5 nicht binden, so dass es in diesen Regionen weniger Schnitte gibt. Durch die Sequenzierung aller DNA-Fragmente und die Analyse der Positionen der beobachteten Schnitte ist es möglich herauszufinden, welche Teile des Genoms die meisten Schnitte aufweisen und somit am besten zugänglich sind. Diese Information hat einen großen Einfluss auf Untersuchungen zur Steuerung der Genexpression als Reaktion auf Signale in der Zellumgebung.
Die klassische Analyse von ATAC-seq-Daten erlaubt jedoch keine Vorhersage, wo Transkriptionsfaktoren direkt binden, sondern zeigt nur potenzielle offene Regionen an, in denen sie binden könnten. Andere Techniken können die Bindung von Transkriptionsfaktoren identifizieren, sind aber sehr aufwändig und erfordern große Mengen an Zellmaterial, die oft nicht zur Verfügung stehen. Daher ist die Untersuchung der Bindung von Transkriptionsfaktoren, insbesondere für mehr als einen gleichzeitig, keine leichte Aufgabe.
Mit einer computergestützten Analyse lässt sich die Bindung von Transkriptionsfaktoren vorhersagen

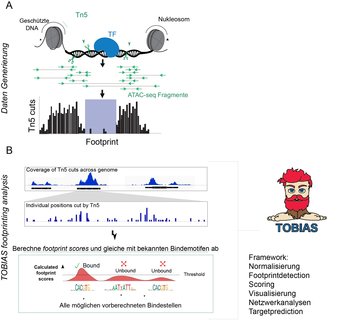
B: Das Programm TOBIAS analysiert die einzelnen Schnittpositionen von TN5 und sucht nach Bereichen mit Signalverringerungen und berechnet einen Footprinting-Score. Dieser wird mit vorhergesagten Bindestellen von Transkriptionsfaktoren verrechnet und dadurch gebundene und ungebundene Bindestellen vorhergesagt. Die Bindestellen werden dann mit umliegenden Genen assoziiert und lassen Rückschlüsse auf deren Regulation zu.
Um diese Probleme zu umgehen, haben wir einen Analyseansatz entwickelt, der vorhandene ATAC-seq-Daten nutzt, um vorherzusagen, wo Transkriptionsfaktoren im Genom binden. Das entwickelte Programm haben wir TOBIAS genannt, was für "Transcription factor Occupancy prediction By Investigation of ATAC-seq Signal" steht. Die Grundlage unseres Ansatzes ist, dass die aktive Bindung von Transkriptionsfaktoren - genauso wie die von Nukleosomen - die DNA vor dem Schneiden durch Tn5 schützt. Dies bedeutet, dass innerhalb größerer offener Regionen, also aktiver DNA, lokal Transkriptionsfaktoren gebunden sind. Genau an diesen Stellen sind dann weniger Tn5-Schnitte vorhanden als in den umliegenden Regionen - ein Effekt, der als Fußabdruck bezeichnet wird (Abb. 1, A). Daher wird diese Art der Analyse allgemein als Footprinting-Analyse bezeichnet. TOBIAS nutzt die Position der ATAC-seq-Schnitte, um nach Footprints zu suchen und sie mit bekannten Transkriptionsfaktor-Zielen abzugleichen. Letzlich versetzt uns diese Analyse in die Lage, global vorhersagen zu können, welche Transkriptionsfaktoren an der DNA wo gebunden haben (Abb.1, B).
Ein Problem beim Footprinting mit ATAC-seq ist jedoch, dass Tn5 eine Präferenz für bestimmte Sequenzen im Genom hat, was zu einem gewissen Rauschen der Daten führt. Daher wendet TOBIAS eine Korrektur an, um diese störenden Hintergrundsignale zu entfernen. Durch diese sind die Fußabdrücke viel einfacher zu sehen und können sogar unter verschiedenen biologischen Bedingungen miteinander verglichen werden. Neben der Identifizierung der Positionen gebundener Transkriptionsfaktoren bietet TOBIAS auch eine Vielzahl von Möglichkeiten zur Visualisierung der Ergebnisse, die für die Interpretation komplexer Versuchsaufbauten wichtig sind.
Biologische Anwendungen der Fußabdruck-Analysen
Aufgrund der beschriebenen Schwierigkeiten bei der Untersuchung der Transkriptionsfaktorbindung sind die Aktivitätsprofile von Transkriptionsfaktoren für viele Zelltypen bislang wenig erforscht. Insbesondere für seltene Zelltypen, wie z. B. in der frühen Embryonalentwicklung, war es bisher schwierig, konventionelle Techniken anzuwenden. Daher haben wir TOBIAS benutzt, um die Bindung von Transkriptionsfaktoren in solchen Zelltypen zu beschreiben.
Dazu wendeten wir unser Programm auf zuvor veröffentlichte ATAC-seq-Daten aus den frühesten Stadien der menschlichen Embryonalentwicklung an. Dabei fanden wir heraus, dass sich die Bindung von Transkriptionsfaktoren in den ersten Zellteilungen sehr dynamisch verändert und konnten anhand der Fußabdrücke zeigen, in welchen Stadien bestimmte Transkriptionsfaktoren aktiv sind. Insbesondere haben wir Transkriptionsfaktoren identifiziert, die sehr spezifisch für das früheste Entwicklungsstadium - das 2-Zell-Stadium direkt nach der Befruchtung - sind, darunter der Transkriptionsfaktor Dux, ein bekannter Vermittler des 2-Zell-Stadiums. Damit konnten wir zeigen, dass ATAC-seq-Footprinting dynamische Veränderungen in der Bindung von Transkriptionsfaktoren auch in den sehr frühen Stadien der Embryonalentwicklung korrekt identifiziert.
Zusammenfassend lässt sich sagen, dass die Footprinting-Analyse eine Möglichkeit bietet, die Art und Weise zu untersuchen, wie die Chromatin-Zugänglichkeit und die Bindung von Transkriptionsfaktoren bei der Kontrolle der Genexpression funktionieren. Diese Analyse unterstreicht zusätzlich, wie maßgeschneiderte computergestützte Analysen aus bereits vorhandenen Daten verborgene Informationen extrahieren können. Unsere Ergebnisse sind vielversprechend für den Einsatz der Footprinting-Analyse zur Untersuchung von Mechanismen der Transkriptionsfaktorbindung in komplexen Zelltypen.