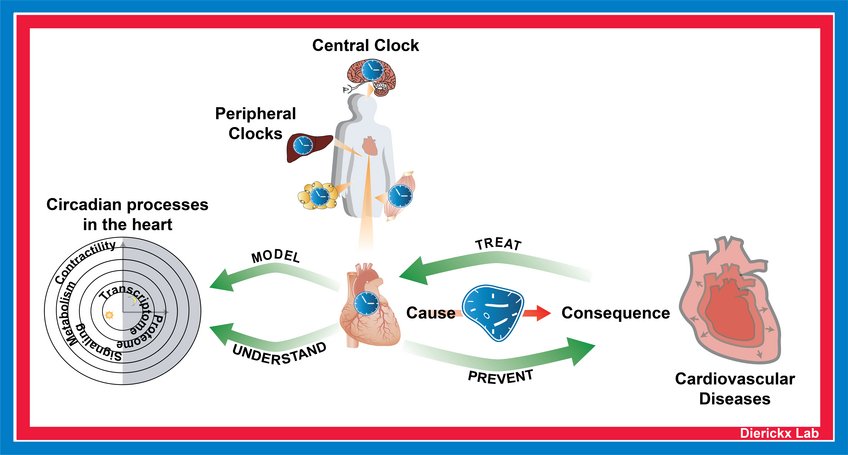
Circadiane Rhythmik des Herzstoffwechsels
Das Dierickx-Labor ist daran interessiert, wie die zirkadiane Uhr rhythmische Prozesse im Herzen steuert. Zirkadiane Rhythmen koordinieren viele Aspekte des Verhaltens und der Physiologie (z. B. Fasten-/Essenszyklen, Schlaf-Wach-Übergänge und Körpertemperatur), um mit der 24-stündigen Erdrotation synchron zu sein. Beim Menschen ist eine Störung dieser Rhythmen mit einem erhöhten Risiko für die Entwicklung von Herz-Kreislauf-Erkrankungen verbunden. Die Unverzichtbarkeit von Uhrenproteinen im Herzen wird mechanistisch durch unsere jüngsten Erkenntnisse veranschaulicht, dass der Verlust von REV-ERB in Kardiomyozyten spezifisch zu dilatativer Kardiomyopathie und vorzeitigem Tod bei Mäusen führt.
Im Mittelpunkt der Projekte des Labors steht ein integrierter Ansatz, der Sequenzierungstechniken der nächsten Generation, Ganzkörperphysiologie-Analysen in speziellen mutierten Mauslinien und die Verwendung von In-vitro-Herzmodellen kombiniert. Unser Ziel ist es, Behandlungsstrategien für kardiometabolische Insuffizienz zu entwickeln, und wir werden Daten von menschlichen Patienten nutzen, um besser zu verstehen, wie die Deregulierung der Uhr zu Herzerkrankungen beiträgt. Das übergeordnete Ziel ist es, zu untersuchen, wie die zirkadiane Uhr und ihre komplizierte Verbindung zu rhythmischen Stoffwechselprogrammen eingesetzt werden können, um Prävention, Diagnose und Behandlung von HF anzugehen.
Wir sind immer auf der Suche nach leidenschaftlichen Menschen, die unser Team verstärken wollen! Erfahrungen mit Tiermodellen, (Stamm-)Zellkulturen und/oder bioinformatischer Analyse von Next-Gen-Daten (z. B. Einzelkern-RNA-seq/ATAC-seq/Cut&Run) wären ein großes Plus.
Informelle Anfragen senden Sie bitte an diese E-Mail-Adresse